Журнал «Актуальная инфектология» Том 9, №5, 2021
Вернуться к номеру
Результати виділення та генотипування вірусів кору, які циркулювали у 2012–2017 роках в Одеській області
Авторы: Гридіна Т.Л. (1), Гончаров В.О. (2), Котлик Л.С. (2), Скопенко А.В. (2), Грузевський О.А. (1), Радкевич К.В. (1)
(1) — Одеський національний медичний університет, м. Одеса, Україна
(2) — ДУ «Одеський обласний лабораторний центр МОЗ України», м. Одеса, Україна
Рубрики: Инфекционные заболевания
Разделы: Клинические исследования
Версия для печати
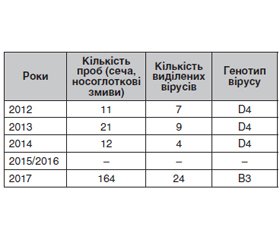
Актуальність. Циркуляція різних штамів вірусу кору тісно пов’язана з регіоном та рівнем захворюваності, оскільки циркулюючі штами можуть змінюватись в період епідемічних спалахів та в міжепідемічні періоди. За даними ВООЗ, найбільш поширеним під час епідемічних спалахів у всьому світі є генотип B3. Тому типування циркулюючих штамів вірусу кору, особливо під час епідемічного спалаху, є важливим процесом, у тому числі й з метою прогнозування розвитку епідемії. Метою даного дослідження була ідентифікація та визначення генотипу циркулюючих в Україні штамів вірусу кору в період 2012–2019 років. Матеріали та методи. Була використана та проаналізована звітна документація ДУ «Одеський обласний лабораторний центр» МОЗУ в Одеській області за 2012–2019 роки щодо дослідження матеріалів від хворих з підозрою на кір з використанням молекулярно-біологічних, генетичних, аналітичних та статистичних підходів. Відповідно до стандартного протоколу ВООЗ для секвенування та проведення філогенетичного аналізу від пацієнтів виділяли циркулюючі штами вірусу кору з використанням спеціальної культури клітин Vero/SLAM, а після культивування з отриманого вірус-вміщуючого матеріалу виділяли РНК вірусу кору та проводили ЗТ-ПЛР. Отриману кДНК відправляли на генотипування, яке проводилось у референс-лабораторії ВООЗ з діагностики кору та краснухи в м. Люксембурзі. Результати. За період 2012–2014 років було виділено 20 штамів вірусу кору з 45 проб (сеча та носоглоткові змиви) від хворих з діагнозом «кір». У 2015–2016 роках виділення вірусу не проводилось через одиничні випадки захворювання. У 2017 році було виділено 24 штами вірусу зі 164 проб. Висновки. Отримані в ДУ «Одеський обласний лабораторний центр» результати свідчать, що у міжепідемічний період 2012–2014 років у регіоні циркулював переважно генотип D4, а починаючи з 2017 року, коли спостерігається підвищення кількості захворілих, пов’язане з новим епідемічним спалахом, на півдні України циркулює переважно генотип В3, генетичної лінії MVs/Kabul.AFG/20.2014/3 B3. Як бачимо, ці дані цілком збігаються з даними, наведеними у літературних джерелах, щодо циркуляції генотипів, які зустрічались у певний час в Європейському регіоні залежно від кількості захворілих.
Background. The circulation of different strains of the measles virus is closely related to the region and the incidence rate since circulating strains can change during epidemic outbreaks and in interepidemic periods. According to the WHO, the B3 strain is most common during outbreaks worldwide. Therefore, typing of circulating strains of measles virus, especially during an epidemic outbreak, is an important process, including for predicting the development of an epidemic. The study was aimed to identify and determine the genotype of measles virus types that circulate in Ukraine during 2012–2019. Materials and methods. Materials of the reporting documentation of the State Institution “Odessa Regional Laboratory Center of the Ministry of Health of Ukraine” in the Odessa region during 2012–2019 were used and analyzed. Materials from patients with suspected measles were used for molecular biological, genetic, analytical, and statistical approaches investigation. Following the standard WHO protocol for sequencing and phylogenetic analysis, circulating measles virus strains were isolated from the patient in a special culture of Vero/SLAM cells. Measles virus RNA was isolated from the resulting virus-containing material after cultivation and RT-PCR was performed. The resulting cDNA was sent for genotyping, which was carried out at the WHO reference laboratory for the diagnosis of measles and rubella in Luxembourg (WHO RRL). Results. Twenty strains of measles virus from 45 samples (urine and nasopharyngeal swabs) from patients diagnosed with measles were isolated during 2012–2014. Virus isolation was not carried out in 2015–2016 due to isolated cases of the disease. Twenty-four virus strains from 164 samples were isolated in 2017. Conclusions. The results obtained at the State Institution “Odessa Regional Laboratory Center” demonstrated that during the interepidemic period of 2012–2014, the D4 genotype circulated in the region. But since 2017, when there was an increas of cases associated with a new epidemic outbreak, B3, genetic line MVs/Kabul.AFG/20.2014/3 B3 mainly circulates in the region of southern Ukraine. As you can see, these data completely coincide with the data about circulating genotypes that were found at a certain time in the European Region, according to the data from the literature.
кір; лабораторна діагностика; полімеразна ланцюгова реакція; виділення циркулюючих штамів вірусу; генотипування
measles; laboratory diagnostics; polymerase chain reaction; isolation of circulating virus strains; genotyping
Вступ
Матеріали та методи
/30.jpg)
Результати
/30_2.jpg)
Обговорення
Висновки
- Rota P.A., Moss W.J., Takeda M., de Swart R.L., Thompson K.M., Goodson J.L. Measles. Nat. Rev. Dis. Primers. 2016 Jul 14. 2. 16049. doi: 10.1038/nrdp.2016.49. PMID: 27411684.
- Griffin D.E., Lin W.H.W. and Nelson A.N. Understanding the causes and consequences of measles virus persistence [version 1; peer review: 2 approved]. F1000Research 2018. 7(F1000 Faculty Rev). 237. Режим доступу: https://doi.org/10.12688/f1000research.12094.1
- Гридіна Т.Л. Кір: збудник, особливості патогенезу, можливі ускладнення, профілактика. Одеський медичний журнал. 2018. № 6. С. 69-74. Режим доступу: http://nbuv.gov.ua/UJRN/Omj_2018_6_16
- Griffin D.E. Measles virus. In: Knipe D.M., Howley P.M., editors. Fields Virology. Volume 6. Wolters Kluwer. Lippincott Williams & Wilkins; Philadelphia, PA, USA, 2013. Р. 1042-1069.
- Naim H.Y. Measles virus: A pathogen, vaccine, and a vector. Human Vaccines & Immunotherapeutics. 2015. № 11(1). Р. 21-26. Available from: http://doi.org/10.4161/hv.34298
- Griffin D.E. Measles Vaccine. Viral Immunology. 2018. Аhead of print. Available from: http://doi.org/10.1089/vim.2017.0143
- Holzmann H., Hengel H., Tenbusch M., Doerr H.W. Millennium Development Goals. United Nations. Eradication of measles: Remaining challenges. Med. Microbiol. Immunol. 2016. 205. Р. 201-208.
- Benecke O., DeYoung S.E. Anti-Vaccine Decision-Making and Measles Resurgence in the United States. Glob. Pediatr. Health. 2019 Jul 24. 6. 2333794X19862949. doi: 10.1177/2333794X19862949. PMID: 31384629; PMCID: PMC6657116.
- Cutts F.T., Lessler J., Metcalf C.J. Measles elimination: Progress, challenges and implications for rubella control: Expert Rev. Vaccines. 2013. 12. Р. 917-932. WHO. Global Measles and Rubella Strategic Plan 2012-2020 [Internet]. 2012 [cited 2017 Mar 5]. Available from: http://apps.who.int/iris/bitstream/10665/44855/1/9789241503396_eng.pdf.
- World Health Organization. Measles fact sheet no. 286. Updated February 2019. Available from: http://www.who.int/mediacentre/factsheets/fs286/en/
- Ristić M., Milošević V., Medić S., et al. Sero-epidemiological study in prediction of the risk groups for measles outbreaks in Vojvodina, Serbia. PLoS ONE. 2019. 14(5). e0216219. Available from: https://doi.org/10.1371/journal.pone.0216219
- Ledd C., Cinà D., Garozzo S.F., et al. Vaccine-preventable disease in healthcare workers in Sicily (Italy): seroprevalence against measles. Future Microbiology. 2019. Published Online: 12 Jun 2019. Available from: https://doi.org/10.2217/fmb-2018-0263
- Гончаров В.О., Котлик Л.С., Скопенко А.В., Грузевський О.А., Гридіна Т.Л. Епідемічні показники щодо кору в Одеській області. Актуальна інфектологія. 2019. 7(2). Available from: http://www.mif-ua.com/archive/article/47270. doi: http://dx.doi.org/10.22141/2312-413x.7.2.2019.161152
- 8th meeting of the European RVC, 12-14 June 2019. https://www.euro.who.int/__data/assets/pdf_file/0019/413236/8th-RVC-Report.pdf.
- WHO. Global Measles and Rubella Strategic Plan 2012–2020 [Internet]. 2012 [cited 2017 Mar 5]. Available from: http://apps.who.int/iris/bitstream/10665/44855/1/9789241503396_eng.pdf
- Kalinichenko, S.V., Melentyeva, K.V., Toryanik, I.I., et al. Genetic monitoring of endemic measles virus circulation in European countries. Annals of mechnikov institute. 2020. 2. Р. 57-70. Available from: http://doi.org/10.5281/zenodo.3885184
- Laksono B.M., de Vries R.D., McQuaid S., et al. Measles Virus Host Invasion and Pathogenesis. Viruses. 2016. № 8(8). P. 210. Available from: doi: 10.3390/v8080210
- Randall R.E., Griffin D.E. Within host RNA virus persistence: mechanisms and consequences. Curr. Opin. Virol. 2017. № 23. Р. 35-42.
- WHO. Policy statement on data sharing by WHO in the context of public health emergencies (as of 13 April 2016). Wkly Epidemiol. Rec. 2016. 91(18). P. 237-40.
- Lekana-Douki S.E., Sir-Ondo-Enguier P.N., Banga-Mve-Ella O., et al. Epidemiology and molecular characterization of the re-emerging measles virus among children and adults in the Haut-Ogooue, Gabon BMC. Infectious Diseases. 2019. 19. P. 90.
- Zaidi S.S.Z., Hameed A., Rana M.S., et al. Identification of measles virus genotype B3 associated with outbreaks in Islamabad, Pakistan, 2013–2015. Journal of Infection and Public Health. 2018. 11 (4). P. 540-545. Available from: https://doi.org/10.1016/j.jiph.2017.10.011.
- Deng Xiuying, Hu Ying, Lu Peishan, Zhou Ming-hao, Guo Hongxiong. The first outbreak of measles virus caused by imported genotype D8 in Jiangsu province of China. Brazilian Journal of Infectious Diseases. 2019. 23(1). P. 66-69. Epub May 20, 2019. Available from: https://dx.doi.org/10.1016/j.bjid.2019.02.003.
- Wong C.H., Chuang S.K., Lam W.H., Lam H.Y., Lam T.S., Ho L.M.R., et al. Investigation and control of a measles outbreak at the Hong Kong International Airport, 2019. Western Pac. Surveill Response J. 2020. 10(2). Available from: doi: 10.5365/wpsar.2019.10.2.007
- Cheng W.Y., Wang H.C., Wu H.S., Liu, M.T. Measles surveillance in Taiwan, 2012–2014: changing epidemiology, immune response, and circulating genotypes. J. Med. Virol. 2016. 88. Р. 746-753. Available from: https://doi.org/10.1002/jmv.24392
- Hartoyo E., Wiyatno A., Jaya U.A., Ma’roef C.N., Monagin C., Myint K.S., Safari D. Occurrence of measles genotype D8 during a 2014 outbreak in Banjarmasin, South Kalimantan, Indonesia Hartoyo. International Journal of Infectious Diseases. 2016. 54. Р. 1-3. Available from: DOI: https://doi.org/10.1016/j.ijid.2016.10.029
- Miyoshi M., Komagome R., Yamaguchi H., et al. Detection of Measles Virus Genotypes B3, D4, D5, D8, and H1 in the Surveillance System in Hokkaido, Japan, 2006-2015, the Last Decade toward the Elimination. Japanese Journal of Infectious Diseases. 2017. 70(3). P. 317-319. Available from: https://doi.org/10.7883/yoken.JJID.2016.253
- Komabayashi K., Seto J., Tanaka S., et al. The largest measles outbreak, including 38 modified measles and 22 typical measles cases in its elimination era in Yamagata, Japan, 2017. Jpn. J. Infect. Dis. 2018. 71. Р. 413-418. doi: 10.7883/yoken.JJID.2018.083
- Seki F., Miyoshi M., Ikeda T., et al. Nationwide Molecular Epidemiology of Measles Virus in Japan Between 2008 and 2017. Front. Microbiol. 2019. 10. 1470. doi: 10.3389/fmicb.2019.01470
- Richard J.-L., Mäusezahl M., Basler S., Eckert N. Approaching measles elimination in Switzerland: changing epidemiology 2007–2018. Swiss Med. Wkly. 2019. 149. w20102. doi: http://dx.doi.org/10.4414/smw.2019.20102
- Savicka O., Kolupajeva T., Aniscenko A., et al. Measles virus genotypes circulating in Latvia, 2011–2018. Available from: https://express.converia.de/frontend/index.php?page_id=10122&additions_conferenceschedule_action=detail&additions_conferenceschedule_controller=paperList&pid=23127&hash=eabff76cae018d35c35277d7eab285ae796e3199bbc67a09fa898a545396c84b
- Эпидемиологическая оценка отдельных заболеваний, предотвращаемых вакцинацией. Эпидемиологическая справка ВОЗ. 2018. № 1. Режим доступа: http://www.euro.who.int/__data/assets/pdf_file/0004/371434/epibrief-1-2018-rus.pdf?ua=1.